Translation of mRNA
단백질 수준에서 생성, 유전자 발현 조절
; 세포 내에서 생물학적 기능을 담당하는 것의 거의 대부분은 단백질
; transcription 수준에서 유전자 발현 조절이 가장 크게/많이 나타나기는 하지만, 유전자 발현 조절은 translation 단계에서도 일어남
; translation 작업은 진화적으로 잘 보존된 process > 원핵과 진핵에서 상당히 유사한 process를 통해 진행 (보편적 기작)
; 해독 공장으로서의 ribosome / mRNA와 단백질간의 adaptor 역할을 하는 tRNA
tRNA
; 어떻게 tRNA가 mRNA 형태로 복제된 유전 정보를 궁극적인 player인 단백질로(아미노산으로) 만들어내는 역할을 하는가? > 구조에 대한 궁금증
; 끝에 아미노산을 붙일 수 있는 고유한 능력이 있음(효소 aminoacyl tRNA synthetase의 촉매기능을 통해 covalent bond 형성)
; 70~80 nucleotide long, 복잡한 이차 구조 형성(cloverleaf)
; tRNA synthetase는 특정 아미노산 / tRNA의 anticodon 을 각각 기질로 삼아 작용
aminoacyl tRNA synthetase (2step working)
- aminoacyl AMP 형성; 아미노산을 activation된 형태로; ATP를 가수분해하며 AMP를 꼬리에 붙여줌
- AMP가 charging된 아미노산은 동일한 효소에 의해 또다른 기질인 tRNA를 만나 tRNA의 3′ end의 amino acid attachment site에 붙음
; tRNA synthetase가 가지고 있는 특이성이 중요 > 61개(64 – stop codon)의 codon에 대해 20종류의 아미노산 matching이 이루어져야 함
; matching 작업을 하는 것이 tRNA synthetase의 역할
; 아미노산 하나당 평균 3개 정도의 코돈 부여
; tRNA synthetase는 기질로 하나의 아미노산 + 그에 matching되는 anticodon을 가지는 평균 3개 정도의 tRNA 할당
; codon의 유전 정보를 아미노산 정보로 매개시켜주는 역할
codon과 anticodon의 base pairing
; 3rd codon position (3번째 염기에서 일어나는 코돈과 안티코돈간의 base pairing은 loose하다)
; codon의 첫번째/두번째는 specific한 base pairing = Watson-Crick base pairing (A-T/G-C)
; 세번째는 훨씬 더 loose한 base pairing > wobble hypothesis (거품과 같은, loose한 형태의 base pairing)
; non Watson-Crick 형태의 base pairing이 빈번히 세 번째 포지션에서 일어난다
ex. G-C가 일어나기도 하지만, 어떤 tRNA는 codon position에 U가 있을 때 anticodon에 G가 주어지기도 함
The ribosome
ribosome = protein factory
; 단백질과 RNA 공존 – RNP complex (RNA+protein)
; ribosome을 구성하는 RNA를 rRNA, ribosome을 구성하는 단백질을 ribosomal protein
; 원핵과 진핵의 ribosome 구조가 큰 윤곽 면에서 비슷
; sedimentation coefficient(침강계수)가 70; 원핵 70S = 50S + 30S / 진핵 80S = 60S(28S, 5.8S, 5S)+ 40S(18S)
모든 RNA는 세포 내에서 secondary structure 형성 (base들로 구성되어 있기 때문에 자기들끼리 base pairing해서, stem loop 구조)
; 리보솜에서 가장 핵심이 되는 protein transferase activity(하나의 아미노산을 다른 아미노산에 붙여주는 역할) 하는 애가 단백질이 아닌 RNA
; 기존에 발견된 모든 효소는 단백질이었는데, 리보솜 내에서는 효소 역할을 수행하는 게 단백질이 아니라 RNA
; 단백질은 Rnp complex를 형성해서 구조적인 역할 > RNA가 구체적으로 효소 기능을 수행할 수 있는 3차원적 구조 형성할 수 있도록 도와줌
; RNA의 catalytic activity 을 가장 먼저 발견한 사례는 self-replicating; RNA self-replcation (RNA의 복제 현상 관찰)
; RNA 복제 역할 하는 애를 찾기 위해 단백질을 다 죽이기; 그래도 replication 일어남 > ribozyme : RNA도 효소 기능이 있다!
RNA의 pepetide bond formation activity를 확인한 결정적인 실험
; tRNA를 puromycine이라는 가짜 아미노산에 붙여줘서 f-Met-puro이 만들어지는 과정을 관찰
; 단백질 제거 시에는(protein extraction) f-Met-puro가 생겼는데, RNA 제거 시에는 생기지 않음
; 리보솜 내에서 f-Met-puro를 만드는 주체가 RNA다
The process of translation
Translation initiation
; scanning을 통한 small subunit의 positioning + large subunit이 오면서 complex 형성
; initiation codon을 인식하는 anticodon을 가지는 methionine/tRNA가 eIF(eukaryote initiation factor)2에 의해 40S subunit으로 옴
; 40S subunit은 이미 다른 eIF들과 assembly되어 있음
; methionine/tRNA와 40S subunit이 binding, complex가 mRNA에 charging되면 initiation
; mRNA에도 많은 factor들이 붙어 있음
; eIF4E는 m7G cap를 specific하게 인식하여 m7G에 binding
; eIF4E가 eIF4G를 통해 PABP(poly A binding protein)와 interaction > mRNA 5’와 3’는 가까워져서 직접 interaction하는 구조 형성
; mRNA는 접혀서 3차원적 구조 형성
; mRNA에 40S subunit이 가서 붙으면 ATP를 가수분해하며 scanning 시작
; AUG를 만나면 멈추고, 60S subunit이 eIF5B에 의해 assembly
; 60S가 assembly되며 40S와 mRNA에 있던 initiation factor들은 다 떨어져 나감 > ribosome+tRNA의 initiation complex 형성
대부분의 진핵세포는 scanning hypothesis에 따라 translation initiation을 하는데, 간혹 다른 방식으로 시작되기도 함; IRESs
; IRES는 5′ UTR을 구성하는 sequence의 일부로, 40S subunit이 scanning하는 대신 중간으로 들어와서 binding할 수 있는 기능 제공
; IRES가 가지고 있는 information이 직접적으로 40S에 제공되기도 / IRES를 인식하는 eIF4G에 의해 제공되기도(eIF4A와 complex를 이뤄서 IRES 인식)
; scanning indepent하게 AUG start codon을 인식하는 경우도 있다
Translation elongation
; polypeptide chain 합성하는 elongation stage
; eukaryote elongation factor = eEF
; 60S ribosomal subunit에 3개의 chamber 존재
; P site에 메싸이오닌을 가지는 tRNA가 들어옴
; 두 번째 아미노산을 가지는 tRNA부터는 A site로 들어오기 시작함 by GTP binding form eEF1α
; 60S ribosomal subunit이 가지고 있는 촉매작용에 의해 peptide bond 형성, P site에 있는 아미노산이 A site의 아미노산 쪽으로 옮겨감
; P site의 tRNA를 E site로 밀어내고, A site의 tRNA를 P site로 옮김 (translocation, 상대적인 이동) by GTP binding form eEF2
; 두 번째 아미노산부터는 코돈에 여러 tRNA가 왔다갔다, 코돈을 인식해서 매치되는 아미노산을 가지는 tRNA가 붙는 게 아니라 들어갔다 안 맞으면 나가고의 반복 > 적합한 애가 들어오면 그때 안정적으로 머무는 것
; 오래 머물고 있으면 base-pairing이 정확한지 추가적으로 확인하는 기능을 40S subunit이 수행, 수행하는 부분을 decoding center라 함
; termination codon이 나타날 때까지 계속 진행
; elongation이 진행되기 위해서는 안정적인 GTP binding form eEF1α의 공급이 있어야 함
; GDP binding form을 다시 GTP binding form으로 바꿔주는 eEF1βγ
; eEF1βγ처럼 eEF들 중 직접적으로 직접 ribosome에서 기능하기보다는 recycling 기능해 주는 애들도 있다
Translation termination
; A site에 termination codon이 오면 match되는 tRNA가 없고 아무것도 안정적으로 머물지 못함(나갔다 들어왔다만 반복)
; release factor protein이 A site에 들어오면 안정적으로 머물 수 있음
; 아미노산이 없어 peptide bond formation이 진행되지 못하므로 폴리펩타이드가 자연스럽게 ribosome을 떠나게 됨(release factor가 붙어있는 상태에서 peptide transferase activity가 작용하면 polypeptide의 growth는 일어나지 않고 release가 대신 진행)
; polypeptide의 release에 따라 ribosome의 subunit들이 모두 분해됨
Regulation of Translation
; initiation stage에서 조절되는 게 가장 efficient (energy cost 측면에서) > 가장 많은 조절 사례 관찰 가능
translational repressor
; ferritin (철을 내부에 보관하고 있다가 필요하면 내보내는 단백질, iron buffer)
; ferritin은 세포 내 철이 어느 정도 있을 때 만들어져야 함 > 세포 내 철 함량을 보고 ferritin 단백질 합성 조절
; 철 함량이 일정수준 이상일 때는 정상적으로 합성 진행
; 철이 없을 때, IRP(iron regulatory protein)가 5′ UTR의 IRE(iron response element)에 가서 붙음. IRP는 철의 함량에 따라 activation이 조절되는 단백질. 40S binding 막아서 initiation complex 만들어지는 걸 막음
3′ UTR을 통한 translation 조절
; poly A tail에 binding하는 translational repressor
; translational repressor가 3′ UTR에 붙어서 eIF4E가 eIF4G와 interaction하는 걸 막음 > PABP-eIF4G-eIF4E의 interaction을 막아 initiation complex 형성 억제
RNA interference
; 세포에 없던 RNA가 갑자기 만들어지거나, 세포에 원래 있던 RNA이지만 그 RNA 양이 over-expression 되었을 때 오히려 그 RNA를 통해 표현되는 생물학적 기능이 없어져버리는 현상
; RNA의 over expression 시 RNA와 단백질이 없어지는 현상(RNA에 의한 유전자 발현 간섭 현상)으로부터 발견
; RNAi를 유발하는 원인들로 small RNAs (regulatory noncoding RNA 중 작은 애들) 확인
; 대표적으로 두 종류 siRNAs / miRNAs (크기 21-25bp, 21-22bp)의 small RNA들이 RNAi를 일으키는 주범
; 둘 사이의 구분은 biogenesis (생성된 origin에 따라), 최종적으로 만들어진 형태는 매우 유사하나 origin이 다름
; siRNA는 생물체 내에서 여러 이유에 의해 우연히 형성되는 double-stranded RNA 부분이 dicer(RNA 분해효소)에 의해 잘려서 형성
; miRNA는 이것을 encoding하는 개별 유전자가 따로 있음. RNA pol2에 의해 gene에서 mRNA 비슷한 게 만들어지고, 그것이 접혀서 만들어진 double-stranded된 stem-loop 구조가 dicer에 의해 잘려서 형성
; miRNA 형성 시 Drosha > Dicer를 거치는 double step cleavage는 포유류에서만, 다른 모든 거에서는 dicer에 의해서만
; siRNA나 miRNA는 유용한 정보를 가지고 있는 powerful한 RNA 조각들이지만 RISC 하에서만 생물학적 기능 가능 (RNA 상태로는 기능할 수 없음) > protein complex의 중요성
; RISC에 들어간 small RNA는 target mRNA를 만날 기회를 가짐
; 둘 사이의 perfect match > mRNA를 자름, 파괴 (드묾)
; 둘 사이의 nearly perfect match > mRNA로부터 단백질로의 해독 저해
; 궁극적으로 silencing (유전자 발현 저해)
; 식물에서는 perfect match되어 mRNA degradation이 많고(nearly perfect match 많음), mammal에서는 inhibition of translation이 많음(mis match 많음)
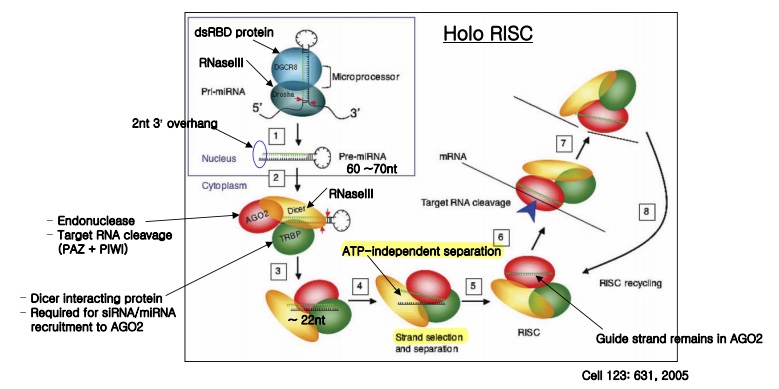
; miRNA를 encoding하는 유전자로부터 RNA 합성되면 (by RNA pol2) 자기들끼리 base pairing > stem loop 구조 형성 (전형적인 RNA의 2차 구조, 자기들끼리 base pairing할수록 열역학적으로 유리한 구조)
; base pairing된 부분은 stem / pairing되지 못하고 남은 부분이 loop
; drosha-DGCR8가 microprocessor complex를 형성하여 stem 구조를 인식 > drosha가 pri-miRNA를 잘라서 pre-miRNA 형성 (drosha가 RNase 3 activity를 가지는 catalytic component, drosha의 기능을 돕는 double strand RNA binding protein인 DGCR8)
; 나와서 RISC complex를 만나서 dicer가 pre-miRNA를 한번 더 자름 (loop 구조 자르기, stem 구조만 남음)
; double strand 상태인 mature miRNA에서 한 strand를 버림(strand separation, ATP 가수분해 없이), 남은 애를 guide strand라고 부름, 얘는 AGO2 단백질에 charging
; RISC의 AGO2는 guide strand와 서열정보가 맞는 target mRNA를 찾아주고(base pairing), mRNA를 자름 > 단백질로 해독될 수 없도록 잘려서 파괴됨 > RNA interference
; 포유동물에서는 핵에서 pri-miRNA가 잘려서 pre-miRNA가 핵 밖으로 나옴
miRNA는 대부분 유전자 발현을 저해하지만, 때로는 오히려 촉진하는 경우도 있음(activation 유발)
; 자르는 과정은 이해했는데, nearly perfect match일 때 어떻게 translation을 initiation stage에서 저해하는지 아직도 100% 정확히 설명하지 못함
; 가능성 중 하나 MRE(miRNA responsive element, miRNA가 작동하는 염기서열 3’UTR 부분)에 miRNA가 matching
; Ago2가 가지고 있는 단백질의 MC domain이 eIF4E의 특정 domain과 비슷하기 때문에 m7G cap에 경쟁적으로 붙을 수 있음
initiation factor의 활성 조절
- eIF2
; eIF2를 통해 tRNA가 공급되어 P site로 위치
; 그런데 growth factor의 생장이 멈춘다면, eIF2와 eIF2B의 인산화 > eIF2B는 GDP binding eIF2를 GTP binding form으로 바꿔주므로 translation initiation 불가능
; phosphorylation을 통한 protein 활설 조절 - eIF4E
; 세포 배양 시 growth factor가 없으면 단백질을 계속 만들면 안 됨 > 생장 멈춰야 함
; eIF4E에 binding하는 4E-binding protein > 얘가 eIF4E에 붙으면 eIF4E가 eIF4G와 binding하지 못해서 initiation complex 형성 안 됨
; growth factor가 들어가면 4E-BP가 phosphorylated > eIF4E에 달라붙지 못해 정상적으로 initiation complex 형성
Protein Folding and Processing
Chaperones and Protein Folding
polypeptide chain 만들어진 후에도 이후 step이 있음 > protein 활성 조절
; 1차 구조를 가진 polypeptide chain이 적합한 3차원적 구조를 형성하는 과정. protein folding
; protein folding은 원핵/진핵 둘 다 나타남 > 진화적으로 얼마나 중요했는지 알 수 있음
; folding에 필요한 기본적인 정보는 그 아미노산 서열에 대부분 들어 있음 (어떤 3차 구조로 갈지 대강은 예측 가능)
; 추가적인 folding 도우미 chaperones, 이제 막 합성이 시작되어 아직 합성이 진행되고 있는 polypeptide chain에 binding하여 정확한 3차원 구조 형성을 도와줌
; polypeptide chain이 target site로 이동하는 중에도 chaperone들은 이것들이 misfolding되어 aggregation되지 않도록 유지시켜줌
Chaperones
; Hsp70, chaperonin 두 종류가 제일 중요
; protein folding task가 커지는 때 = 세포 온도 조건이 급격히 바뀔 때(단백질 변성)
; 그러한 상황에서 단백질들이 정확한 folding 상태를 유지하도록 도와주는 애들; heat-shock proteins = Hsp (heat shock시 많이 형성되는 얘가 chaperon이라는 걸 나중에 알게 됨)
; chaperonin이라는 거대한 protein chamber 안에서 folding하기도 함 (chaperonin은 protein folding을 도와주기에 적합한 내부 환경을 가지고 있음)
Protein Misfolding Diseases
정확한 3차원적 구조를 가진 단백질이 생성되지 않으면, 독성이 강한 새로운 기능을 수행하기도 함 (질병)
; 단백질이 만들어지지 않아서 생기는 질병이 아니고, 단백질이 엉뚱한 구조로 변형되면서 protein aggregation되면서 생기는 질병
; 정상적인 3차원적 구조를 가진 단백질이 변성이 되면서 변성된 형태의 단백질들이 누적되어 형성되는 것을 총칭하여 amyloid 계열이라 부름
; amyloid β의 누적이 알츠하이머 병으로 된다는 게 아직까지 일반적인 의견
제일 유명한 게 prion
; 정상적 conformation은 PrP c
; 변형된 구조 PrP sc가 정상 구조 단백질과 붙어서 정상 단백질의 비정상적 구조 변형을 유발 (prion propagation)
; 비정상 프라이온의 비율이 일정 수준을 넘어가면 질병에 걸림
; prion-like protein도 많음 (amyloid β도 비슷하게 만들어지는 것으로 이해)
Enzymes that Catalyze Protein Folding
PDI
; 많은 protein들에 보편적으로 사용되는 protein folding 효소
; disulfide bridge (covalent bond) 형성 > 단백질의 activity나 function에까지 영향 미치기도 함, 정확한 3차원적 구조 유도하는 데에 중요
Peptidyl prolyl isomerase
; 프롤린이 존재할 때 옆에 있는 peptide bond의 conformation을 바꿔줌 (cis-trans간 교체)
; 인접한 polypeptide chain의 구조에까지 영향 (단백질의 overall structure에 영향 > 정확한 protein folding을 이끌기도, 저해하기도)
Protein Cleavage
; 때에 따라 final conformation을 얻기 위해 protein cleavage / proteolysis (단백질 분해)가 동반되어야 함
ex 1. insulin 단백질의 maturation 과정
; translation된 preproinsulin (precursor form)
; 앞쪽에는 ER targeting을 돕는 signal sequence / B peptide / A peptide / 두 펩타이드 주변에 connecting pepetide
; signal sequence의 제거, A-B peptide간의 disulfide bridge 형성 (proinsulin)
; connecting polypeptide 제거 (matured insulin)
; functional한 insulin이 겪는 두 단계의 proteolysis
ex 2. HIV protein의 maturation 과정
; AIDS를 유발하는 single-strand RNA virus인 HIV
; reverse-transcriptase에 의해(HIV 연구에서 처음 발견됨) cell로 infection된 후 DNA로 genome 전환
; HIV가 encoding하는 유전자 중 하나인 protease, virus 껍데기를 구성하는 단백질을 최종적으로 maturation 시킴
; protease가 정확히 작동하지 않으면 virus particle이 만들어지지 않아 증폭 실패
; AIDS 치료를 위해 HIV protease / reverse-transcriptase 의 inhibition 연구 (요즘은 drug mix로 효과 잘 보고 있음)
Attatchment of Carbohydrates and Lipids
Glycosylation
; 만들어진 단백질(실은 nascent polypeptide chain)에 carbohydrate라는 당 polymer (sugar residue) 붙이기 > 이에 따라 단백질의 활성/위치 조절
단백질에 carbohydrate가 어느 position에 붙느냐에 따라 크게 두 그룹으로 나눔
; N-linked glycoprotein (아미노산 side chain의 nitrogen에 sugar residue가 붙음 ex. asparagine)
; O-linked glycoprotein (아미노산 side chain의 oxygen에 sugar residue가 붙음, hydroxyl group을 가지고 있을 때 hydroxyl group 내의 산소에 붙는 경우 많음. ex. serine)
glycoprotein이 가장 많이 발견되는 곳
; 대부분 secretion (만들어진 후 세포 밖으로 배출) / secretory pathway를 따라 plasma membrane에 최종적으로 end up
; sugar group은 plasma membrane에 anchoring될 때 세포 바깥쪽으로 노출되어 있음
Attachment of Lipids
; 단백질이 sugar residue를 통해 lipid와 결합
; membrane 구성 요소 중 inositol 같은 lipid가 있을 때 여러 종류의 sugar 붙음 (glycolipid=lipid+carbohydrate)
; glycolipid를 특정 단백질이 만나게 되면 단백질의 C terminus가 당에 붙음
; 결국 protein anchored (ex. GPI)
; cytosol에서 만들어진 단백질이 더 이상 자유롭지 않게 됨, membrane 주변에만 맴돌게 됨 (peripheral-membrane protein)
; 단백질도 주로 세포 바깥쪽으로 노출
- plasma membrane을 구성하는 단백질들 중 세포질 안쪽을 향하고 있는 단백질에 lipid가 붙는 경우 (주로 signal transduction에 있어 중요한 역할을 함)
- N-myristoylation
; 탄소14개 가지고 있는 지방산(myristic acid)이 N-terminal을 구성하는 글라이신 residue에 가서 붙음
; polypeptide N terminal에는 흔히 starting codon Met이 있는데, N-myristoylation이 일어나기 위해 먼저 methionine이 제거됨
; Met이 제거되고 다음 아미노산이 Gly일 때 흔히 글라이신에 myristic acid가 가서 붙는 일이 일어난다 - Prenylation
; protein C terminus의 Cys-A-A-X에서 시스테인이 가지고 있는 side chain의 sulfur에 가서 특정 종류의 lipids가 붙는 경우
; 세포의 growth/differentiation과 연관, Ras oncogene에 의해 encoding되는 단백질이 대표적으로 prenylation에 의해 조절되는 단백질 - Palmitoylation
; 16 carbon fatty acid가 polypeptide chain의 internal cys residue sulfur에 붙는 경우
Regulation of Protein Function
추가적인 protein function 조절
- small molecule이 protein에 붙었다 떨어졌다 하며 protein의 기능이 조절되는 사례
- 공유결합을 통해 protein에 phosphate group과 같은 특정한 chemical group이 붙고 떨어지며 하며 protein의 기능 조절
- 많은 protein들은 혼자가 아니라 protein complex로 기능 > partner subunit과 잘 만나고 헤어짐에 따라 protein의 기능 조절
- 만들어진 protein의 분해도 중요 (특정 시점의 protein 총량과 관련)
1. regulation by small molecules
; small molecule은 protein에 non-covalent bond (hydrogen bond/hydrophobic interaction/ionic interaction 등)을 통해 붙었다 떨어졌다 함
; 특히 protein function이 조절될 때 allosteric regulation이 일어남 (small molecule이 binding하는 regulatory site와 실제 protein의 function을 표현하는 catalytic site가 다르다)
; 궁극적으로는 catalytic site에 의해 표현되는 protein function이 조절된다
; small molecule은 activator/inhibitor로 작용함
; non-covalent bond이기 때문에 촉매 없이도 binding이 반복(readily reversible, 빠르게 가역적으로 조절)
ex. Ras oncogene protein은 GDP와 binding했을 때 inactive form, GTP와 binding하면 단백질 구조가 바뀌며 protein 활성이 생김
2. protein phosphorylation and other modifications
; protein kinase; 온갖 종류의 단백질에 phosphate group을 붙이는 효소를 총칭, 안에 subgroup 많음 (어디에 phosphate group을 붙이느냐에 따라, ser/thr kinase, tyr kinase, …)
; serine/threonine이 가지고 있는 OH group에 가서 .. / tyrosine의 ring 구조에 가서 … => substrate specificity
; protein phosphatase; 단백질에 이미 붙은 phosphate group을 제거하는 효소를 총칭
; 둘은 서로 경쟁적 관계, 어떤 것의 활성이 높은지에 따라 target protein에 phosphate가 붙기도 떨어지기도 함, 이것들의 총량과 활성을 조절
ex. phosphorylation에 의해 조절되는 대표적인 경우 – glycogen phosphorylase
glycogen; 동물이 당을 저장할 때 사용하는 polymer, 간에 저장하다가 당 결핍 신호가 오면 glycogen을 분해시켜 glucose 형태로 만들어서 에너지원으로 사용
저장물질을 에너지원으로 전환하는 것이 glycogen phosphorylase; 얘의 활성 조절이 신체 내 당 level 조절에 중요
이것의 활성 조절은 single step으로 일어나지 않음, 이 자신도 phosphorylation에 의해 조절 (phosphorylation cascade, signal amplification 각 step을 거치며 signal이 커짐, 적은 양의 hormone으로 많은 양의 glucose 생성 가능)
궁극적으로 glucose level 조절에 있어 cAMP를 얼마나 활성화시키느냐가 중요, 이것을 조절하는 것은 epinephrine 같은 hormone
ex. phosphorylation 이외의 mechanism
NO(nitric oxide); 혈액 내에 gas 형태의 hormone, 이게 protein의 internal Cys residue에 붙는 걸 nitrosylation이라 함, 붙고 떨어지고에 따라 on/off
이외에도 다양한 chemical group이 붙고 떨어짐에 따라 효소나 단백질의 활성이 조절된다
3. protein-protein interaction
multi protein이 참가하여 protein complex를 형성하는 경우
ex. cAMP-dependent protein kinase
; 평소에 regulatory subunit 2개 + catalytic subunit 2개로 구성된 complex 형성
; hormone 신호에 의해 세포 내 cAMP level이 높아지면 cAMP가 regulatory subunit에 붙음
; regulatory subunit의 구조가 바뀌며 더 이상 catalytic subunit과 interaction하지 못하게 됨
; catalytic subunit이 풀려나며 활성화됨
; regulatory subunit과 catalytic subunit의 interaction이 궁극적으로 catalytic subunit으로 표현됨
4. protein degradation
; input과 output을 통해 궁극적으로 조절해야 할 것은 steady state level, 어떤 특정 물질이 세포 내에서 안정적으로 유지되는 농도
; 분해 진행 방식 두 가지, lysosomal proteolysis는 엄청 거대한 스케일에서 일어남, 여기서는 ubiquitin-proteasome pathway 다룸
Ubiquitin-Proteasome pathway
; ubiquitin을 붙여주는 part / ubiquitin이 붙은 것들을 분해하는 part
; proteasome은 poly ubiquitin chain(death signal)이 붙은 애들만 분해 by ATP 가수분해
; 세포는 아주 선별적으로 poly ubiquitin chain을 붙임; 세 종류 효소들이 어느 단백질에 붙일지 결정 (E1, E2, E3)
; E1은 ATP를 사용해서 ubiquitin을 자기 몸에 붙임 (ubiquitin activation)
; activate된 유비퀴틴을 E1에서 E2로 E2가 넘겨받음
; E2는 E3가 결정한 단백질에 ubiquitin을 붙임 (E3는 자기 몸에 유비퀴틴을 붙이지는 않음)
; E3가 기질 특이성 결정! (substrate specificity ubiquitylation)
; monoubiquitinylation >(반복) polyubiquitnylation > poly ubiquitin chain 형성 (mono ubiquitin은 death signal로 기능하지 못함)
; ubiquitin은 아무데나 붙지 않고 E1, E2, substrate protein의 lysine residue에만 붙음 (lysine-specific)
; E3는 제한된 substrate만 인식 가능 > 한 세포 내에는 수많은 degradation target이 되어야 하는 단백질 존재
; 한 세포에 수백 개의 다른 종류의 E3 존재 > 모든 target 인식
cf. E1, E2는 적은 숫자 존재
; poly ubiquitin chain이 반드시 death signal로만 작용하지는 않음. endocytosis의 target signal으로 작용하기도 함
; histone 단백질에 대해서는 polyubiquitinylation을 통해 degradation 될 수도 있지만, 그 경우는 드묾
; 히스톤 단백질을 구성하는 여러 lysine residue에 ubiquitin이 하나 붙으면(histone monoubiquitinylation) death signal 대신 히스톤 단백질의 conformation을 조절 > chromatin 구조 조절 (histone code로 작용) or DNA repair로 기능하기도 함
; ubiquitin보다는 작은 SUMO
; poly sumorylation이 일어나지는 않음
; 그러나 protein에 가서 ubiquitin처럼 붙었다 떨어졌다는 많이 일어남
; degradation signal로 작용 x
; 다양한 방법으로 protein의 활성을 조절 (enzyme 활성 조절 / protein localization 결정 / histone sumorylation 시 histone code로 기능)
Ubiquitination에 의한 cell cycle 조절 by cyclin-dependent kinase
; phosphorylation을 시키며 cell cycle의 on/off or speed를 조절하는 kinase, kinase의 활성 조절하는 건 cyclin
; cyclin은 interphase-specific하게 합성 유도
; Cdk와 결합 > cdk 활성 살아남 > cell cycle progress 진행
; cyclin B에 ubiquitination이 서서히 진행되기 시작
; 세포 내 cyclin B level 점점 감소
; cyclin B 없는 Cdk (불활성)
; cell cycle은 더 이상 진행되지 못함
; cell cycle과 cyclin level이 비례함을 알게 됨
; 세포분열이 진행되며 cyclin level이 주기적으로 왔다갔다
; 왔다갔다에는 만들어지는 것보다 분해되는 게 더 중요함을 알게 됨 (ubiquitination-dependent degradation)