Gene Regulation in E.coli
; 최초의 전사 단계에서의 유전자 발현 조절 모델, 가장 단순하면서도 보편적인 시스템 갖춤
; 주변 환경의 변화에 빠르게 대응할 수 있는 방식, 자신의 대사 능력을 빠르게 바꾸는 박테리아
lac operon
; lactose가 주변에 있을 때 사용하기 위해 디자인된 유전자 발현 조절 모델
; lactose는 6탄당 두 개가 붙어 있는 12탄당 형태, 분해되면 galatose/glucose로 e.coli가 실제 사용할 수 있는 형태로 바뀜
; lactose를 분해하여 에너지원으로 사용하는 데 필요한 유전자들이 lac mRNA에 존재
; 유전자 z, y, a는 operator에 의해 공통적으로 조절되며 i는 별도로 조절
; promoter는 RNA polymerase와 general transcription factor가 부착하는 DNA sequence
; i가 발현되면 lac repressor protein 생성
; lactose가 없을 때 lac repressor는 operator sequence에 달라붙음 > RNA pol이 promoter에 달라붙을 수 없음 (promoter와 operator의 sequence 중복으로) > z, y, a의 transcription 억제
; lactose가 없다면 lactose metabolism에 필요한 유전자들을 발현시킬 필요가 없기 때문에, lac repressor를 통한 shutdown은 효율적임
; lactose가 존재하면 lactose는 lac repressor와 결합함으로써 z, y, a가 발현되도록 유도
; lactose binding lac repressor는 구조가 변형되어 operator에 붙지 못함 > free promoter에 RNA pol 결합 > 전사 > z, y, a의 trasncription > lactose metabolism에 사용되어 e.coli가 lactose를 energy source로 사용
∴repressor를 활용하여 유전자 발현을 shutdown/open (negative control)
positive control 방식도 사용
; metabolism에 가장 활용되기 좋은 형태인 glucose
; glucose가 있으면 무조건 glucose를 쓰고, glucose가 없으면 차선으로 lactose 등을 사용 by positive control
; glucose는 해당과정을 거치며 5탄당 α-ketoglutarate로 변화 > α-ketoglutarate는 adenylyl cyclase를 inhibit
; adenylyl cyclase는 ATP를 cAMP로 바꿔주고, cAMP는 CAP에 binding하여 CAP의 활성 촉진 > 활성화된 CAP은 DNA에 붙어서 RNA pol α subunit과 interaction > RNA pol의 promoter 결합 도와줌 > 유전자 발현 촉진 (positive regulation)
; glucose가 있으면 lactose가 있더라도 glucose를 먼저 사용 (preferentially utilized) > lactose를 에너지원으로 사용하는 데 필요한 유전자들의 발현을 촉진하지 않음
; glucose가 없으면 adenylyl cyclase 활성 > cAMP(activator)를 이용한 유전자 발현 촉진 > glucose 대신 lactose와 같은 alternative sugar의 metabolism 활성
∴ glucose 유무에 따라 positive regulation pathway가 먼저 작동하고, 뒤이어 lactose 유무에 따라 repressor 기능이 저해되거나 촉진되는 pathway가 작동
Transcription Factors in Eukaryotes
진핵생물 특이적인 유전자 발현 조절; 핵에 위치한 DNA, enhancer, epigenetics
enhancer; RNA polymerase와 general transcription factor가 붙었다 떨어졌다 하며 유전자 발현을 조절하는 promoter뿐 아니라,
promoter의 기능을 한층 더 강화시키는 enhancer가 등장
epigenetics; 진핵생물의 DNA가 naked 상태인 대신 chromatin 구조에 embedding되어 있어서
enhancer
; 원핵생물의 promoter 중 가장 simple한 구조를 가지는 herpes virus의 promoter
; 진핵생물은 아니지만, 진핵생물 세포 안으로 침투하여 유전자 발현을 구사하는 애기 때문에 진핵생물이 가지는 promoter와 공통적인 특성을 가지고 있음(진핵생물이 가지고 있는 유전자 발현 조절 체계를 거의 그대로 가지고 있음)
; promoter에는 element 3개 이상 (TATA box, CAAT box, GC box) > 전체가 promoter
; SV40의 enhancer에는 promoter 외에도 뭔가 다른 게 유전자의 발현을 조절(새로운 sequence element) > enhancer라 이름 붙임
; enhancer는 promoter의 기능을 향상시키는 역할(on/off뿐 아니라)
promoter의 정의 – RNA pol / RNA pol과 complex를 이뤄서 함께 작용하는 general transcription factor가 달라붙는 부위
; enhancer는 RNA pol이 붙는 데가 아니면서도 promoter의 기능을 강화시킴
enhancer test 기법; 유전자 앞에 이런 element가 있는 것으로 의심
PCR, northern blot까지 하지 않더라도(mRNA의 양을 직접 측정하지 않더라도) 눈으로 유전자의 발현 정도를 쉽게 관찰할 수 있는 애들을 reporter gene이라 함
reporter gene 앞에다 의심되는 DNA sequence element 집어넣기, 세포에 집어넣어서 관찰
promoter 기능을 강화시켜주는 enhancer 확인
promoter 기능 강화 이외의 enhancer의 추가적인 특성
; enhancer 없이 promoter만 있으면 유전자 발현이 낮은 수준에서 > basal transcription
; enhancer 추가하면 transcription 활성이 강화됨 > 많은 RNA copy 생성
; enhancer는 promoter 바로 앞에 위치할 때만 일하는 게 아님 (엄청 많이 떨어져 있어도 강화)
; promoter와의 거리와는 independent (distance independent)
; promoter 반대쪽에 뒤에 가 있어도 똑같이 활성, 위치와도 무관 (position independent)
; 방향 뒤집어도 괜찮음 (orientation independent)
; 세 가지 특성, independency
이 모든 것이 DNA가 3차원적 구조로 존재하여 loop를 만들 수 있기 때문
; loop를 통해 enhancer가 promoter와 가까워질 수 있음 (3차원적으로는)
; loop를 통해 세 가지 특성 모두 설명
; 1차원적 구조로는 멀리 떨어져 보이지만 실제 세포 내에서는 아주 가까울 수 있는 것
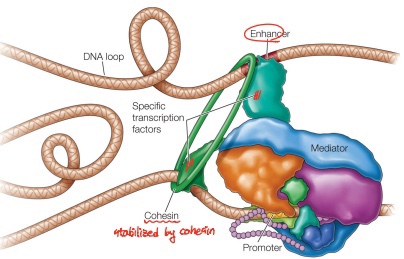
; loop 형성 시 loop 형성 도와주는 애들 > cohesin, CTCF > 특정한 preference를 가지고 loop 형성(위치에 비례하여 loop 만들어지고 이런 건 아님)
; dimer로 기능하는 CTCF
; cohesin 혼자서는 안 됨, cohesin과 CTCF는 늘 붙어다님(사진 잘못됨)
; cohesin-CTCF complex는 loop 형성 촉진 > enhancer와 promoter의 distance 가깝게 해 줌 (이것만 해주는 애는 아니고, 원래는 그냥 온갖 loop 형성)
; promoter에는 RNA pol과 general transcription factor가 붙고, enhancer에는 또 다른 종류의 transcription factor가 붙음
; promoter에 붙은 RNA pol/transcription factor와 enhancer에 붙은 transcription factor 간의 가교 역할을 하는 것이 mediator(protein complex), 직접 DNA에 붙는 것 아님
; loop 형성을 통해 promoter와 enhancer가 physically 가까워지면 각각에 붙는 여러 단백질들에 mediator가 가교 역할을 하여 이것들간의 실제 physical interaction을 도움
; enhancer는 세포 타입 dependent한 특성이 있음 > 어떤 세포 타입에서만 보이는 기능이 있음 > enhancer 발견의 어려움
cis element (cis-acting regulatory sequence)
; DNA 염기서열 상의 어떠한 유전자의 발현을 조절하는 sequence element
; cis element란 DNA 상에 존재하는 염기 서열 그 자체, 자체로서 유전자 염기 서열
; cis element는 혼자 작동하지 않고, 거기에 DNA binding protein(ex. transcription factor)이 붙어서 작용
; cis element에 붙는 DNA binding protein을 trans element라 부름
; 유전자 발현 조절은 cis element와 trans element에 의해 이루어진다
; trans element가 특정한 cis element를 찾아서 붙은 후 궁극적으로 RNA pol의 활성을 촉진, positive regulation
; promoter, enhancer도 cis element
; promoter, enhancer에 붙는 transcription factor(activator, repressor, …)이 trans element
; mediator는 trans element가 아님(co-repressor or co-activator) *직접 DNA에 binding하는 애들에 가서 붙는 것들
cis/trans element를 찾기 위한 실험 기법들
enhancer의 cis부터 찾기(염기 서열 자체부터) > cis에 붙는 binding protein을 찾기(cis에 매칭되는 trans 찾기)
EMSA
아가로스 젤에 DNA 포함 solution 넣고 전기영동
; negative charge인 DNA는 시간에 따라 +극 쪽으로 이동 (size에 따라)
; DNA에 단백질이 붙었다면 size가 커져서 느리게 이동(electrophoretic-mobility shift)
; 온갖 단백질 다 넣어줌 > 이 DNA는 DNA-binding protein이 붙을 수 있는 애구나(cis 찾기) > 단백질 특정 > trans element임을 증명
; 원하는 DNA 잘 잘라올 수 있음, mutation을 통해 정밀하게 증명 가능(EMSA의 반복을 통해). specific한 narrow down 가능, cis의 핵심적인 염기가 뭐인지까지 가능
; 아주 구체적으로 acting하는 cis element가 뭔지 알 수 있음 > 이용하여 promoter, enhancer 등을 구체적으로 define할 수 있었음
; 단백질을 모르는 상태에서도 시작할 수 있고, 의심가는 단백질이 있을 때 할 수도 있음
chip-seq
; 빠르게, 대량으로 assay
; 세포나 조직으로부터 chromatin fraction를 뽑아냄
; 뽑기 전에 포름알데하이드를 처리하여 수소결합/이온결합/소수결합과 같은 비공유결합으로 형성된 DNA/transcription factor complex를 공유결합 complex로 전환 > 세포/조직의 fixing (안정적인 complex 상태가 되어 분리되지 않음)
; 잘게 쪼개기 위해 sonication (강도, 파장의 크기에 따라 쪼개는 크기 조절 가능) > 보통 500bp 내외로 쪼갬, 필요에 따라서는 200bp 정도의 mono nucleosome 크기로
; test하고자 하는 DNA binding protein이 있다면, 그에 부착하는 항체를 넣어줌
; 여러 DNA binding protein이 붙어 있는 chromatin fraction 중 specific하게 항체가 인식할 수 있는 단백질을 가지고 있는 chromatin fraction에 항체가 붙음
; spin down > 원하는 단백질이 붙은 항체, 단백질이 붙어 있는 DNA가 한꺼번에 다 같이 면역침강됨
; reverse cross-linking > 화학처리를 통해 단백질과 항체가 떨어져 나감, 면역침강된 fraction 내에 DNA만 남음
; DNA에 대해서는 PCR / DNA sequencing으로 어떤 DNA가 있는지 알 수 있게 됨
; 내가 검증하고자 하는 염기서열에 가서 진짜 이 단백질이 붙는지 특정한 sequence에 대해서만 테스트할 수 있기도 함
- 내가 test하고자 하는 단백질이 DNA에 가서 직접 붙는지
- 직접 붙는다면 붙는 sequence가 어떤 sequence인지
알 수 있음 (EMSA와 비슷한 걸 얻어낼 수 있음)
EMSA는 DNA를 먼저 알고 거기에 붙는 단백질을 찾는 과정이라면
chip은 단백질을 먼저 알고 단백질이 붙는 DNA seqeunce를 찾는 과정
; 궁극적으로 실험이 주는 데이터의 성격은 비슷하다
modified chip = chip-seq
; sequence를 small scale 대신 large scale로 하면 세포 내에 내가 테스트하고자 하는 단백질이 붙는 전체 sequence를 모두 밝혀낼 수 있음
; genome (DNA 염기서열의 총합) 내에 target sequence를 한꺼번에 밝혀낼 수 있음
Purification of Sp1 by DNA-affinity chromatography
DNA & DNA binding protein간의 interaction 관계에 기초하여 사람들이 처음으로 특정한 DNA(cis element)에 binding하는 단백질(transcription factor, trans element)을 찾아낸 실험
; 전체 세포를 깨서 핵만 얻어냄
; 핵에 있는 단백질을 gel에 사이즈별로 분리시킴
; column에 한번 통과시킨 후 남아있는 단백질들만 loading
; column에 한번 더 통과시킨 후 남은 단백질들 loading
; promoter element 중 하나인 GC box를 가지고 있는 DNA를 agarose bead라는 커다란 당에 붙여줌 > 얘네를 쌓아서 mini column 만듦
; 여기에 핵에서 뽑은 단백질을 부으면 GC box DNA에 붙는 애들은 column에 남아 있게 됨 / 붙지 않는 애들은 빠져나감
; 진짜 붙는 애들만 남게 해 주기 위해 washing해 줌 > 아주 specific하게 붙는 애들만 남게 해 줌
; high salt buffer를 처리해 DNA binding protein과 DNA sequence간의 비공유결합 interaction이 깨짐
; 궁극적으로 붙어 있던 단백질만 column 밖으로 나옴 > 단백질 동정
; GC box sequence를 붙여줬을 때 거기에 specific하게 binding하는 단백질이 Sp1임을 밝힘
; affinity chromatography > column을 구성하는 물질과 column 위에 loading되는 물질간의 친화력을 사용 (친화력이 높은 애들만 이후에 정제)
Transcription regulatory proteins
transcription 자체를 수행하는 건 RNA pol
RNA pol에 작용하여 전사 촉진; transcriptional activator
; 그 자체가 DNA binding protein, 또 다른 activation domain이 있고 아주 다양한 방법으로 이게 전사 촉진
ex. mediator를 통해 RNA pol complex와 interaction / 염색질 구조를 변형시키는 애들을 데려와서 열린 염색질 구조로 변화시킴
RNA pol에 작용하여 전사 언제; transcriptional repressor
- 또 다른 DNA binding protein으로 경쟁적으로 작용 (activator의 binding 방해)
- repressor는 own dna binding sequence를 찾아서 붙음, 그리고 repression domain을 통해 여러 방법으로 작용
ex. mediator를 통해서 RNA pol의 활성을 저해 / chromatin을 닫힌 구조로*activator/repressor는 직접 DNA에 붙어서 작용, coactivator/corepresser은 activator/repressor에 의해 recruit되어 들어와서 작용
Regulation of elongation
RNA pol이 promoter에 붙어 일을 시작 = transcriptional initiation = firing
firing되었다 해서 그 다음 transcription이 작동적으로 되지는 않음, 지속되기 위한 step으로서 elongation
; RNA pol 2가 가지고 있는 CTD(C terminal domain)
*단백질이 translation해서 제일 처음 만들어지는 부분이 N terminal domain / 마지막 완성되는 부분이 C terminal domain
; RNA pol 2의 CTD는 hepta-peptide repeat (똑같은 서열을 가지고 있는 아미노산 7개가 반복)
; 7개의 반복된 아미노산을 1~7로 numbering했을 때 2/5번째에 있는 serine
; serine의 phosphorylation이 왔다갔다, 이게 transcription 조절하는 데에 중요한 역할을 함
; 5번째 serine이 인산화되면 firing 시작 > initiation (인산화되지 않으면 polymerase가 promoter에 붙어있을 뿐, RNA transcription을 안 함)
; phosphorylation은 TFIIH protein kinase가 인을 붙여줘서 됨
; 50bp 정도 만들고 stall in
; stall in 상태에서 elongation 상태로 바뀌며 process가 계속 진행되기 위해서는 serine 2 phosphorylation을 시켜줘야 함
; P-TEFb가 인산화시켜줌 > NELF는 떨어져나가고 DSIF는 인산화된 채로 남아 있으면서 RNA pol의 성격이 바뀜 > elongation
*NELF와 DSIF는 negative regulatory factors
∴ RNA pol 2에 있는 CTD의 hepta peptide repeat에 어떤 종류의 serine phosphorylation(serine 5 / serine 2)이 되는지에 따라 initiation/elongation 되기도 한다
Chromatin and Epigenetics
prokaryote의 DNA는 almost naked – transcription factor가 붙었다 떨어졌다를 빠르게 반복할 수 있음
진핵생물의 DNA는 핵 내에서도 coated되어 있음, coated된 그 구조를 chromatin이라 함
- chromatin; DNA에 histone 단백질/비 histone 단백질/RNA/transcription factor … 가 덕지덕지 붙어 있는 구조
puffing된 구조; decondensation되어 부피가 증가해 있는 염색질 구조 > 이 부분이 유전자들은 발현 잘 됨 (열린 염색질 구조)
DNA는 먼저 histone 단백질을 감고 있음(7/4바퀴, 147bp) – nucleosome
nucleosome – linker DNA – … ; nucleosome+linker DNA는 200bp
histone에는 core histone(DNA가 감고 있는) / linker histone(histone H1, linker DNA에 달라붙어 있는)
core histone에는 histone 4개가 참여(histone H3, H4, H2A, H2B) .. 각각 2 copy 씩 > 총 8 copy가 형성한 구; core histone
histone의 N-terminal domain을 구성하는 아미노산들에는 아주 다양한 chemical modification이 일어남
; histone acetylation – acetyl group이 histone 단백질의 lysine에 가서 붙음 (modification 중 첫번째 발견) – 공유결합성 modification
; 진핵생물의 DNA를 coat하고 있는 histone 단백질들의 chemical modification에 따라 염색질 구조가 닫히고 열림을 발견 – 유전자 발현의 억제/촉진
; HAT(Histone acetyl transferase)에 의한 acetylation은 염색질을 여는 방향으로 작용 > 전사 촉진
; HDAC(Histone deacetylase)에 의해 normal lysine으로 돌아가면 염색질 닫힘 > 전사 억제
∴ 단순하게 RNA t/f의 활성이 아닌, DNA가 감싸고 있는 히스톤 단백질들에 어떠한 종류의 modifcation이 일어나는지에 따라 RNA pol / tf의 binding efficiency가 영향을 받아 transcription이 잘 되기도 하고, 안 되기도 한다(2차 조절)
이러한 종류의 유전자 발현 조절은 진핵생물에서만 독특하게 찾아볼 수 있음(epigenetics)
lysine에 일어나는 acetylation을 최초로 증명한 실험
; yeast의 Gcn5가 transcriptional coactivator로 작용한다는 것을 포닥 때 확인, 그 메커니즘을 알지 못했음
; 짚신벌레를 이용해서 실제 histone 단백질의 acetylation을 시킬 수 있는 단백질이 있다는 것을 In-gel activity assay로 알아냄 > 그 단백질이 Gcn5의 상동체(homolog)였음
histone code (cf. genetic code)
; 유전자 발현 조절에 있어 on/off switch로 작용하는 histone 상의 chemical modification
적어도 12가지 종류의 chemical modification이 밝혀짐 (유전자 발현 조절, 염색질 구조 조절)
; met/ac는 lysine에만 일어남 (Arg에서도 met일어날 수 있음)
; phs는 hydroxyl group을 가지고 있는 serine, threonine, tyrosine 등에 일어남
; 이런 것들은 주로 histone core의 N-terminal tail쪽에서 일어남
; nucleosome 바깥쪽으로 돌출되어 있어서 modification을 유발하는 효소들 ex. HAT/HDAC가 쉽게 접근
nucleosome의 어떤 위치에 어떤 패턴의 modification이 나타나냐에 따라 유전자 발현 조절이 달라지기도 함
; acetylation은 무조건 전사 촉진
; lysine mono-met(Kme1)는 전사 억제, di/tri-met(Kme2/Kme3)는 전사 촉진
; histone H3의 4번째 아미노산 lysine (H3K4) 에의 met3일 때 전사 촉진
; H3K27met3는 전사 억제
; histone code는 무한대의 조합으로 chemical modification 결과 나타남
NFR(Nucleosome free region) /NDR
; 200bp마다 하나씩 있다가 통째로 nucleosome이 빠져 있는 데가 있음
; 주로 promoter, enhancer 쪽에 (아주 빈번하게 사용되는 promoter/enhancer 중에는 NFR인 경우가 있음)
; 그 부분의 cis element가 막강한 regulatory role을 할 수 있게 됨
NFR/NDR 현상의 수행
; chromatin remodeling factor
; histone의 특정한 chemical modification을 유발하여 chromatin 구조를 변화시키지는 않음
; 원래 있던 nucleosome을 빼내기도, 넣기도, 위치 이동시키기도 (ATP를 에너지원으로 사용하며) > NFR 생성, 제거
; nucleosome의 position/density 변화를 통해 chromatin 구조 바꾸기
histone modification/chromatin remodelling에 의해 한 세포에서 일어난 chromatin 구조의 변화가 세포분열 이후 딸세포에서도 여전히 유지; epigenetic inheritance
; 단순하게 transcription activity를 on/off 시킬 뿐 아니라 다음 세대에서도 유전자가 사용되거나 사용되지 않은 패턴을 결정
; 유전자의 존재 유/무 이상으로(mendel 유전학), 유전자가 사용될 수 없는 염색질 구조에 있으면 그 유전자는 발현되지 않을 수 있다
; 유전자 사용 유/무를 결정하는 epigenetics by chromatin 구조
; 그 chromatin 구조가 한번 형성되면 딸세포로 계속 전해질 때가 있다
; 특정한 histone modification / 특정한 chromatin 구조는 세포분열 이후에도 지속적으로 유지되기도 한다, 특정한 유전자가 계속 사용되거나/사용되지 않도록 결정한다
; 유전자 사용 유무를 염색질 구조를 통해 지속적으로 결정(유전이 가능한 패턴으로)
; met되어 있던 DNA가 replication
; modification된 nucleosome을 절반씩만 나눠가짐
; replication 이후 새로 만들어진 nucleosome이 들어옴
; 새로 들어온 nucleosome도 met됨 (modification pattern 유지)
how?) lysine27의 methylation을 읽을 수 있는 reader 중 하나가 PRC1 + 다른 nucleosome을 methylation 시킬 수 있는 PCR2
한 nucleosome이 met되어 있으면 그걸 읽을 줄 아는 1이 그걸 읽고, 친구를 데리고 옴. 그 친구는 다른 nucleosome에 동일한 met을 시킬 수 있음 > met
; parental nucleosome이 met 되어 있으면 그 met을 새로 들어온 nucleosome에도 copy해서 집어넣음; 동일한 modification pattern 유지
염색질 구조를 결정하는 데 중요한 역할을 하는 것에는 template DNA상에 직접 일어나는 met도 있음
cf. 보통은 cytosine 5번 탄소의 met가 제일 유명(염색질 구조 닫는 쪽, corepressor 기능 수행)
최근에는 adenine 9번 탄소에도 met 일어남
DNA 상의 met은 훨씬 더 쉽게 딸세포에서 유지될 수 있음
; met된 parental DNA가 정보를 줘서 새로 만들어진 DNA 가닥을 methylation시킴
; 그런 효소들이 거의 모든 세포에 존재하여 DNA met은 복제 이후에도 잽싸게 일어남
한번 met되면 계속 유지된다고 생각해 왔는데, DNA met의 역과정도 일어남이 최근에 밝혀짐 (active DNA Demethylation)
; methylation을 지우는 효소가 발견
; 동물에서 그 일을 수행하는 효소를 TET라 부름
; met > hydroxyl methyl > fomil > carboxyl
; fomil이나 carboxyl group을 가지고 있는 변형된 cytosine은 base-excision repair을 통해 아예 잘리고, met되지 않은 새로운 cytosine이 심어짐(DNA repair)
; TET enzyme과 base-excision repair 효소의 작용에 의해 demethylation
cf. passive DNA Demethylation
maintenance of methylation이 작동하지 않으면 세포분열이 지속되면서 methylation된 DNA 농도가 감소하는 것
Genomic Imprinting
; DNA met에 의한 gene repression과 관련
; Parent-of-origin effect가 더 과학적으로 의미 있는 용어
; met되지 않은 egg + met된 sperm (같은 1번 chromosome)
; zygote 상태에서도 met + met x
; zygote로부터 개체가 형성될 때, met된 건 사용되지 않고 met되지 않은 건 사용
; 어느 parent로부터 그 유전자를 물려받았는지에 따라 그 효과가 다르게 나온다
; 동일한 유전자를 물려받았다 할지라도 어떤 유전자는 사용되지 않을 수도 있음. 물려받지 않은 것과 동일한 결과 cf. Mendel genetics
noncoding RNAs
; RNA는 coding RNA와 nonconding RNA가 있음
; DNA가 담고 있는 정보를 단백질로 전달시켜 주는 것이 일반적인 coding RNA의 역할 – mRNA
; coding potential이 없는, 단백질을 만들지 못하는 noncoding RNA (RNA가 최종 형태)
- 단순히 구조적 역할만 하는 structural noncoding RNAs; tRNA, rRNA / snoRNA도 인에서 구조적인 역할 수행
- 생물학적 조절 기능을 하는 regulatory noncoding RNAs; 크기에 따라 분류(200bp 기준)
- small; miRNA, siRNA, piRNA (유전자 발현 조절과 관련)
- long; genome이 실수에 의해 만들어낸 junk라고 생각, 알고보니 생물학적 조절 기능에 참여, lncRNA
Xist lncRNA
; X chromosome이 두 개인 여성의 경우, X chromosome 하나는 barr body 형태로 응축되어(chromatin condensation) 사용되지 않음
(dosage compensation; x chomosome 상에 존재하는 유전자의 사용 빈도를 조절)
; RNA가 scaffold로 작용하여, chromatin을 닫힌 구조로 유발하는 factor들을(DNA methy transferase, ..) 데리고 와서 complex 형성
lncRNA는 scaffold 기능만 하지 않고, 제일 중요한 기능은 DNA/histone의 변형을 유발시키는 epigenetic regulator들을 데리고 오는 것
; DNA/histone modifier는 DNA sequence를 인식해서 binding, 저절로 찾아오는 게 아님. lncRNA가 delivery해주는 것, 데려가서 기능할 수 있도록
; DNA와의 specific base-pairing을 통해 특정한 염기 서열을 찾아감(modifier를 데리고!), recruitment, 효소들을 적재적소에
Epigenetics 정리
; histone 단백질에 covalent modification 유발 (covalent histone modification)
; nucleosome 전체를 빼거나 더하기도, 위치를 바꾸기도 (ATP-dependent remodeling)
; replication 직후에 생긴 canonical histones / 필요에 따라 histone 단백질을 다른 변형된 것으로 바꿔주기도 (histone variant exchange)
; DNA methylation & demethylation – writer, eraser, redaer, effector, …
; 진핵생물은 이러한 많은 조합을 통해 각 세포 타입마다 같은 유전자라 할지라도 동일한 유전자가 존재하는 chromatin 부위의 개폐를 조절 > 세포의 분화